| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | ||||
| 4 | 5 | 6 | 7 | 8 | 9 | 10 |
| 11 | 12 | 13 | 14 | 15 | 16 | 17 |
| 18 | 19 | 20 | 21 | 22 | 23 | 24 |
| 25 | 26 | 27 | 28 | 29 | 30 | 31 |
- fMRI
- Normalise
- abcd
- neurofeedback
- 코드업
- cortical representation
- Realignment
- 파이썬
- matlab
- Word Embedding
- DCCSAE
- Coregistration
- 한정판텀블러
- socioeconomic status
- DMN
- Kernel regression
- 우박수
- Slice timing
- Python
- SPM
- SPM12
- CodeUp
- 약수구하기
- 판다스
- 판다스기초
- cortical mapping
- pandas
- RSFC-based behavioral prediction
- hierarchical clustering analysis
- 광화문텀블러
- Today
- Total
몽발개발
SPM을 사용하여 뇌영상 preprocessing하기 [5]Normalise 본문
이제까지 처리한 데이터는 개인의 데이터이다. 사람마다 다른 뇌구조를 가지고 있으니 이들을 비교해서 활용하기 위해서는 공통점이 필요하다. 그렇기 때문에 개개인의 fMRI 영상을 표준화된 템플릿에 맞추는 과정이 필요한데, 이것이 normalise이다.
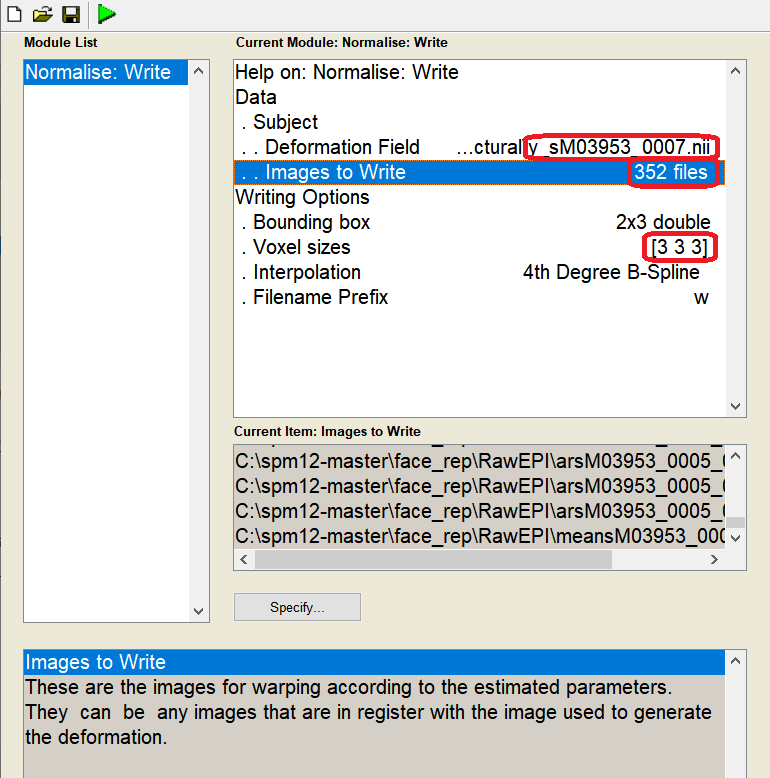
spm 초록창에서 Normalise(Write)을 클릭하면 batch editor가 뜬다.

Data를 누르고 아래 New: Subject를 클릭하면 두 개의 입력값이 추가된다. Deformation Field와 Images to Write인데, Deformation field에는 전 과정인 segment에서 만들어진 deformation이 씌워진 파일 y_sM03953_0007을 선택하고, Images to Write에는 slice_timing까지 처리했던 functional images인 arsM~파일들 351개를 선택한다. 추가적으로 realignment때 만든 mean functional image인 meansM~파일까지 선택하여 총 352개의 파일을 선택하면 된다.
그리고 아래 voxel sizes 에 2 2 2를 클릭해 3 3 3으로 바꾼다. 보통 2 2 2를 사용한다고는 하는데 이번 데이터는 뭔가 다른가보다.(메뉴얼이 시켰어요)
이제 이 batch를 normalise란 이름으로 jobs에 저장후 실행시킨다.
이 과정이 완료되면 RawEPI폴더에 arsM~파일 앞에 w가 추가로 붙은 warsM~파일들이 생기게 된다. 이로써 우린 realignment(완료시 r붙음), slice timing(완료시 a붙음), normalise(완료시 w붙음)를 전부 거친 war~파일들을 얻게 되었다. 이 wars~파일들은 움직임보정, 시간보정, 그 후 표준화된 fMRI 템플릿에 맞춰진 뇌 정보인 것이다.
이제 마지막 과정인 smoothing만 진행하면 preprocessing은 끝이다.
'매트랩(MATLAB) > SPM' 카테고리의 다른 글
| SPM을 사용하여 뇌영상 preprocessing하기 [6]Smoothing (0) | 2020.07.16 |
|---|---|
| SPM을 사용하여 뇌영상 preprocessing하기 [4]Segment (0) | 2020.07.16 |
| SPM을 사용하여 뇌영상 preprocessing하기 [3]Coregistration (1) | 2020.07.16 |
| SPM을 사용하여 뇌영상 preprocessing하기 [2]Slice timing (0) | 2020.07.16 |
| SPM을 사용하여 뇌영상 preprocessing하기 [1]Realignment (1) | 2020.07.16 |